Caso de estudio: Domesticación y fitomejoramiento del trigo
Patrick F. Byrne, Dept. of Soil and Crop Sciences, Colorado State University, Fort Collins, CO 80523 (Patrick.Byrne@colostate.edu)
Contenido
- Introducción
- Los genomas ancestros A y B y la hibridación A x B
- El genoma ancestro D y la hibridación AB x D
- Trigo hexaploide sintético
- Genes útiles de los parientes silvestres del trigo
- Referencias
- Agradecimientos
1. Introducción
La domesticación del trigo (Triticum aestivum L.) desde los pastos silvestres en el Medio Oriente es una historia fascinante que resultó en uno de los cultivos más importantes y ampliamente distribuido en el mundo. Se estima que el trigo proporciona el 20% de la energía en la dieta global y el mismo porcentaje de proteína (WHEAT, 2014). El trigo es ante todo un cultivo de clima templado, pero está ampliamente adaptado a muchos tipos de condiciones de crecimiento, de riego y de temporal, y es especialmente importante como un cultivo básico en condiciones semiáridas. Parte de la adaptabilidad del trigo se debe a que se presenta tanto en variedades de invierno (requieren un periodo de frío de 6 a 8 semanas para desencadenar su reproducción) como en variedades de primavera (no requieren el periodo de frío). Genetistas y fitomejoradores de trigo poseen una larga historia identificando genes útiles en los parientes silvestres del trigo e incorporándolos en las variedades mejoradas.
2. Los genomas ancestros A y B y la hibridación A x B
El trigo común es una especie hexaploide con tres conjuntos de cromosomas similares, pero distintos, denominados genomas A, B y D. Cada conjunto de cromosomas, contiene siete pares de cromosomas. Por lo tanto, el trigo hexaploide contiene 3 genomas x 7 pares = 21 pares o 42 cromosomas totales. Cada genoma se originó de diferentes especies de pastos diploides anuales en la Media Luna Fértil del Medio Oriente (Figura 1).
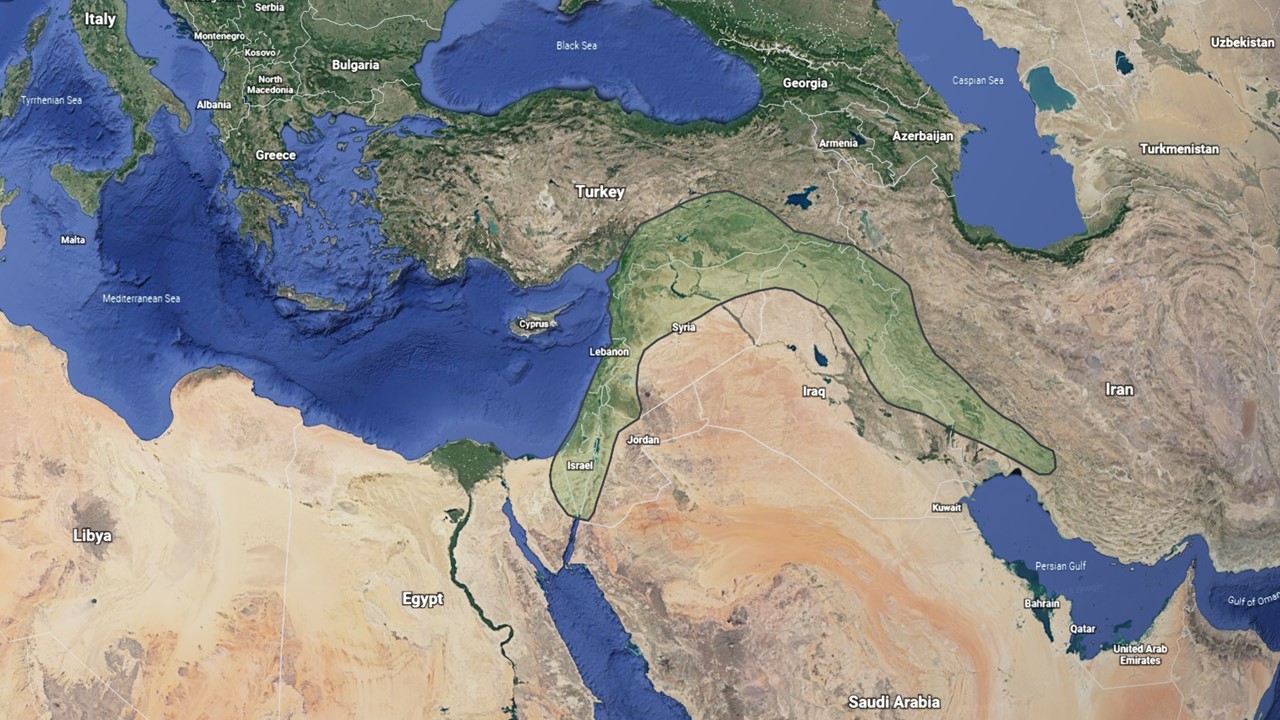 Figura 1. La Media Luna Fértil del Medio Oriente, donde se domesticaron el trigo y muchos otros cultivos.
Figura 1. La Media Luna Fértil del Medio Oriente, donde se domesticaron el trigo y muchos otros cultivos.
 Figura 2. La combinación de los genomas A, B y D originó el trigo harinero, el cual contiene los tres genomas. De izquierda a derecha, genoma A, Triticum urartu; genoma B, Aegilops speltoides ligustica; genoma D, Aegilops tauschii; y genoma A+B+D, Triticum aestivum. Crédito de la foto: Pat Byrne.
Figura 2. La combinación de los genomas A, B y D originó el trigo harinero, el cual contiene los tres genomas. De izquierda a derecha, genoma A, Triticum urartu; genoma B, Aegilops speltoides ligustica; genoma D, Aegilops tauschii; y genoma A+B+D, Triticum aestivum. Crédito de la foto: Pat Byrne.
El ancestro del genoma A del trigo es Triticum urartu. El genoma B se cree que tiene su origen en el pariente cercano Aegilops speltoides. La hibridación entre estos progenitores, hace menos de un millón de años (Marcussen et al., 2014) dio lugar a las especies tetraploides Triticum turgidum ssp. dicoccoides, conocidas como farros silvestres, con genoma AABB. Esta especie silvestre fue domesticada para generar el trigo farro (Triticum turgidum ssp. dicoccum), el cual dio lugar al trigo duro o de pasta (Triticum turgidum ssp. durum). Los trigos farro y duro son tetraploides con un genoma AABB.
Un linaje separado del genoma A condujo a la domesticación del trigo escanda (Triticum monococcum ssp. monococcum), el cual aún crece en partes remotas de Turquía, Italia y España. El genoma de esta especie diploide es usualmente denominado AmAm para distinguirlo del linaje que dio origen al trigo hexaploide.
Video 1. El Dr. Patrick Byrne muestra ejemplos de los ancestros del genoma A, B y D del trigo, describiendo la historia evolutiva del trigo y explica cómo los trigos silvestres son relevantes para el fitomejoramiento moderno del trigo.
Video 2. El Dr. Patrick Byrne destaca ejemplos de rasgos contrastantes en el trigo moderno y sus ancestros silvestres o variedades tradicionales, lo que brinda una visión de la historia del fitomejoramiento del trigo. Los rasgos destacados son altura de la planta, índice de cosecha, acame y ruptura.
Video 3. El Dr. Patrick Byrne muestra espigas cosechadas de los ancestros A, B y D del trigo y explica la estrategia de crear trigo hexaploide sintético para aumentar la diversidad del trigo cultivado.
3. El genoma ancestro D y la hibridación AB x D
El genoma ancestro D del trigo es el pasto de cabra Aegilops tauschii, el cual se produce a lo largo de un amplio rango del Medio Oriente y Asia, desde Siria hasta China. El análisis de ADN del trigo y sus parientes silvestres llevaron a Marcussen et al. (2014) a la conclusión de que el genoma D se originó de una hibridación entre los genomas A y B donadores hace 5.5 millones de años. Mucho después, otra hibridación ocurrió entre el trigo farro (AABB) y Ae. tauschii (DD), dando lugar al trigo hexaploide común o harinero (T. aestivum), con la denominación del genoma AABBDD. Se cree que esta hibridación ocurrió hace solo 8,000 a 10,000 años, probablemente en un área del sur del Mar Caspio hoy en día Irán (Gepts, 2018). El genoma D le proporcionó al trigo la adaptabilidad a las duras condiciones ambientales de Asia Central, así como las proteínas que mejoraron las propiedades de la harina para la elaboración del pan.
Se cree que la hibridación que condujo al trigo común ha involucrado un número muy limitado de plantas Ae. tauschii, creando un efecto cuello de botella donde el genoma D fue incorporado a un cultivo hexaploide. Por lo tanto, genéticamente, el genoma D es el menos diverso de los tres genomas del trigo. La diversidad en la amplia gama de Ae. tauschii casi no se presenta en los trigos de hoy en día y, hasta fechas recientes, no se ha explorado como una fuente de variación útil.
Video 4. El Dr. Patrick Byrne muestra los diversos hábitos de crecimiento del progenitor del genoma D del trigo, Aegilops tauschii, y analiza los procesos utilizados para cruzar Ae. tauschii con trigo harinero.
4. Trigo hexaploide sintético
Una estrategia creativa para manejar la baja diversidad genoma D del trigo es el desarrollo de trigos hexaploides sintéticos (McFadden and Sears, 1946; Trethowan and Mujeeb-Kazi, 2008; Ogbonnaya et al., 2013). Esta estrategia es un intento para recrear la historia evolutiva del trigo mediante cruzamiento intencional del trigo tetraploide (AABB) con Ae. tauschii (DD), pero utilizando un rango mucho más amplio de accesiones de Ae. tauschii en comparación con las que participaron en la naturaleza. Ae. tauschii es una especie atractiva para la introgresión al trigo común, debido a que su genoma se aparea y recombina fácilmente con los cromosomas del genoma D del trigo. El Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT) ha sido un líder en la producción y utilización de hexaploides sintéticos, con más de 1,500 sintéticos desarrollados (Mujeeb-Kazi and Hettel, 1995; Rosyara et al., 2019).
El proceso de desarrollo de trigo hexaploide sintético se representa en la Figura 3. Como el producto inicial de fertilización entre especies tetraploides y diploides es inviable después de unas semanas, en muchos casos se usa un procedimiento de rescate de embriones. Esto involucra la disección del embrión joven y crecerlo en cultivo de tejido para producir una planta triploide (ABD). El número de cromosomas de esta planta es entonces duplicado mediante tratamiento con colchicina para producir plantas AABBDD, las mismas que el trigo común. Otros métodos para incorporar la diversidad de especies silvestres en el trigo incluyen cruzamiento directo de Ae. tauschii o accesiones tetraploides silvestres a trigo hexaploide, así como lo explica Ogbonnaya et al. (2013).
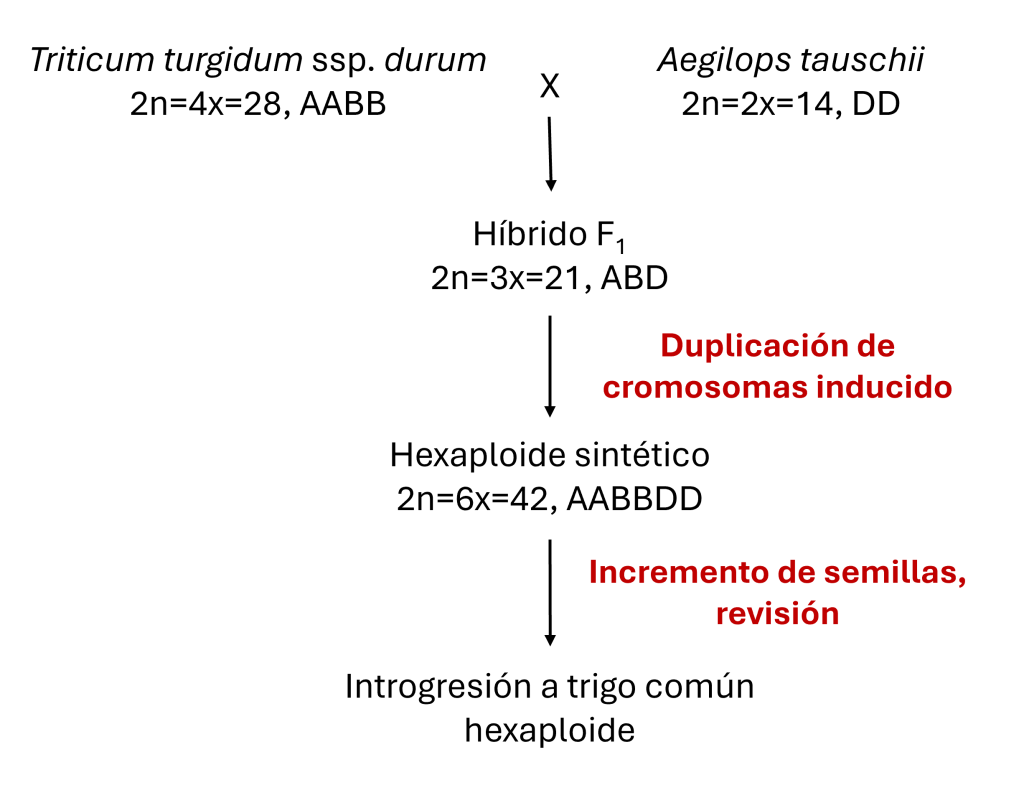 Figura 3. Desarrollo de trigo hexaploide sintético (Ogbonnaya et al. 2013).
Figura 3. Desarrollo de trigo hexaploide sintético (Ogbonnaya et al. 2013).
Los hexaploides sintéticos no son directamente útiles en la producción de trigo porque, en general, no están adaptados al área de crecimiento que se desea y expresan rasgos de los parientes silvestres, tales como una trilla deficiente (por ejemplo, las semillas no se separan fácilmente de la gluma) y el acame excesivo (Figura 3). Por lo tanto, las líneas sintéticas normalmente se cruzan con cultivos de trigo adaptados, después se retrocruzan una o dos veces con el cultivo adaptado, seguido de selección. Esto resulta en líneas mejoradas (a veces llamadas líneas sintéticas derivadas) que tienen genomas adaptados con segmentos de cromosomas hexaploides sintéticos intercalados. Estas líneas crecen y se evalúan como trigo normal con la expectativa de lograr mejoramiento en rasgos cualitativos y cuantitativos debido a la inserción de segmentos hexaploides sintéticos.
 Figura 4. Líneas hexaploides sintéticas de invierno de CIMMYT en crecimiento en Fort Collins, Colorado. Crédito de la foto: Pat Byrne.
Figura 4. Líneas hexaploides sintéticas de invierno de CIMMYT en crecimiento en Fort Collins, Colorado. Crédito de la foto: Pat Byrne.
CIMMYT ha realizado evaluaciones extensas en sus líneas sintéticas derivadas (Dreccer et al., 2007; Lage and Trethowan, 2008; Rosyara et al., 2019). Ogbonnaya et al., 2013 mencionan que, en los 7 años previos a su revisión, de todas las pruebas internacionales de CIMMYT, el 17% de las líneas de trigo fueron líneas sintéticas derivadas, pero ese valor se incrementa a 35% en las pruebas de rendimiento al trigo de la región semiárida. En un estudio de 253 líneas sintéticas derivadas relevantes para el programa de fitomejoramiento y evaluación del CIMMYT, con base en marcadores la estimación promedio de la contribución de Ae. tauschii a esas líneas fue de 17.4% (Rosyara et al., 2019). Más de 80 líneas sintéticas derivadas se han liberado como cultivos, en particular Sokoll y Vorobey (Rosyara et al., 2019).
Las líneas hexaploides sintéticas han demostrado su utilidad como fuente de variación en la tolerancia al estrés abiótico y biótico, así como en rasgos agronómicos y novedosos de calidad de grano. La mejora en su desempeño productivo bajo condiciones de estrés por sequía se debe, al menos en parte, a cambios en su enraizamiento, ya que una mayor proporción de biomasa de raíces se produce a una profundidad mayor (Lopes and Reynolds, 2011; Reynolds et al., 2008).
Otro enfoque para descubrir variaciones útiles en accesiones de Ae. tauschii es desarrollar poblaciones anidadas para mapeo de asociación (NAM), un método descrito en maíz por Yu et al. (2008). Este método fue llevado a cabo por Eric Olson (anteriormente de la Universidad Estatal de Kansas, ahora en la Universidad Estatal de Michigan) mediante cruzamiento directo de siete accesiones de Ae. tauschii en la línea de trigo adaptada de Kansas KS05HW14, seguido de dos retrocruzamientos y varias generaciones de autopolinización. La población resultante de 420 líneas (denominadas como DNAM) se muestra en el Video 5.
Video 5. El Dr. Patrick Byrne muestra parcelas de investigación de diferentes accesiones de trigo con genoma D que se cruzaron con una línea de trigo adaptada de Kansas. Se están estudiando los rasgos de tolerancia a la sequía.
5. Genes útiles de los parientes silvestres del trigo
Los genes de especies de Aegilops y otros parientes silvestres han sido extremadamente importantes para mejorar una serie de rasgos en trigo, en especial la resistencia a enfermedades. Kishii (2019) recopiló genes que han sido identificados o transferidos de Ae. tauschii hacia el trigo. Estos genes incluyen genes de resistencia a la roya de la hoja, roya del tallo, roya lineal, mildiú polvoriento, Septoria tritici, Septoria nodorum, mancha bronceada, nemátodo enquistado, nemátodo nudulador de la raíz, mosca de Hesse, chinche verde, pulgón ruso del trigo, ácaro del rizo del trigo y virus del mosaico de cereales transmitido en el suelo. Otro cuadro en Kishii (2019) recopila genes de especies de Aegilops distintas de Ae. tauschii. Esta lista incluye genes de resistencia a la mancha ocular, roya de la hoja, roya del tallo, roya lineal, mildiú polvoriento, nemátodo enquistado, nemátodo nudulador de la raíz y chinche verde.
El Centro de Recursos Genéticos de Trigo en la Universidad Estatal de Kansas es líder en la colecta, conservación y aprovechamiento de los parientes silvestres del trigo para el mejoramiento del cultivo. La liberación de germoplasma por el Centro incluye líneas mejoradas para la resistencia a enfermedades e insectos, la calidad del pan y otros rasgos.
En el ámbito de la tolerancia al estrés abiótico, Placido et al. (2013) demostraron que la traslocación de un segmento de cromosoma de Agropyron elongatum proporciona al trigo la habilidad de mantener el crecimiento de la raíz bajo condiciones de estrés por humedad.
6. Referencias
Dreccer MF, Borgognone MG, Ogbonnaya FC, Trethowan RM, Winter B. 2007. CIMMYT-selected derived synthetic bread wheats for rainfed environments: Yield evaluation in Mexico and Australia. Field Crops Research 100:218-228.
Gepts P. 2018. The domestication of our food crops. In: Chrispeels MJ, Gepts P (editores). Plants, Genes & Agriculture: Sustainability through Biotechnology. Oxford University Press, Cary, North Carolina.
Kishii M. 2019. An update of recent use of Aegilops species in wheat breeding. Frontiers in Plant Science 10:585.
Lage J, Trethowan RM. 2008. CIMMYT’s use of synthetic hexaploid wheat in breeding for adaptation to rainfed environments globally. Australian Journal of Agricultural Research. 59:461-469.
Lopes MS, Reynolds MP. 2011. Drought adaptive traits and wide adaptation in elite lines derived from resynthesized hexaploid wheat. Crop Science 51:1617-1626.
Marcussen T, Sandve SR, Heier L, Spannagl M, Pfeifer M, International Wheat Genome Sequencing Consortium, Jakobsen KS, Wulff BB, Steuernagel B, Mayer KF, Olsen OA. 2014. Ancient hybridizations among the ancestral genomes of bread wheat. Science 345:1250092.
McFadden ES, Sears ER. 1946. The origin of Triticum spelta and its free—threshing hexaploid relatives. Journal of Heredity. 37:81-89; 107-116.
Mujeeb-Kazi A, Hettel GP, (editors). 1995. Utilizing wild grass biodiversity in wheat improvement: 15 years of wide cross research at CIMMYT. CIMMYT Research Report No. 2. Ciudad de México, México.
Ogbonnaya FC, Abdalla O, Mujeeb‐Kazi A, Kazi AG, Xu SS, Gosman N, Lagudah ES, Bonnett D, Sorrells ME, Tsujimoto H. 2013. Synthetic hexaploids: Harnessing species of the primary gene pool for wheat improvement. Plant Breeding Reviews 37:35-122.
Placido DF, Campbell MT, Folsom JJ, Cui X, Kruger GR, Baenziger PS, Walia H. 2013. Introgression of novel traits from a wild wheat relative improves drought adaptation in wheat. Plant Physiology 161:1806-1819.
Reynolds M, Dreccer F, Trethowan R. 2007. Drought-adaptive traits derived from wheat wild relatives and landraces. Journal of Experimental Botany 58:177-186.
Rosyara U, Kishii M, Payne T, Sansaloni CP, Singh RP, Braun H-J, Dreisigacker S. 2019. Genetic contribution of synthetic hexaploid wheat to CIMMYT’s spring wheat breeding germplasm. Scientific Reports 9:12355.
Trethowan RM, Mujeeb-Kazi A. 2008. Novel germplasm resources for improving environmental stress tolerance of hexaploid wheat. Crop Science 48:1255-1265.
WHEAT. 2014. Wheat: Vital grain of civilization and food security. 2013 Annual Report, CGIAR Research Program on Wheat, Ciudad de México, México.
Yu JM, Holland JB, McMullen MD, Buckler ES. 2008. Genetic design and statistical power of nested association mapping in maize. Genetics 178:539-551.
7. Agradecimientos
Para citar este artículo: Byrne PF. 2024. Caso de estudio: Domesticación y fitomejoramiento del trigo. En: Volk GM, Byrne PF (Eds.) Parientes silvestres de cultivos y su uso en el fitomejoramiento. Colorado: Universidad Estatal de Colorado. [Escribe la fecha de acceso]. Disponible en: https://colostate.pressbooks.pub/cropwildrelativesspanish/chapter/domesticacion-y-fitomejoramiento-del-trigo/
Este módulo de capacitación fue posible, en parte, gracias al financiamiento del USDA-ARS, la Universidad Estatal de Colorado, IICA-PROCINORTE (procinorte.net) y la Agencia de Estados Unidos para el Desarrollo Internacional (USAID). Traducciones a español fueron apoyados por IICA-PROCINORTE.
Editores: Gayle Volk, Katheryn Chen
Traducido: Luis Felipe Guzmán Rodríguez, Andrea Purdy, Stephanie Villanueva, Verónica Villanueva
Camarógrafo: Mike May
Créditos de las fotos del encabezado (de izquierda a derecha): Phil Westra, Pat Byrne, Mike May.
Este proyecto fue financiado en parte por National Academy of Sciences (NAS) [la Academia Nacional de Ciencias] y USAID. Es de notar que las opiniones, los hallazgos, las conclusiones y las recomendaciones expresadas vienen de los autores y no reflejan necesariamente los puntos de vista de USAID ni de NAS.


